Categorical Data Preprocess
- SEX
- SIDO
- HEAR_LEFT
- HEAR_RIGHT
- SMK_STAT_TYPE_CD
- DRK_YN
- HCHK_OE_INSPEC_YN
- CRS_YN
- TTR_YN
SEX
성비
- 남 : 여 = 1.4 : 1
수정
- 남성 : 1
- 여성 : 0
남성이 여성보다 당뇨병일 확률이 근소하게 높다
- 남성 : 0.093142
- 여성 : 0.054500
결측치 없음
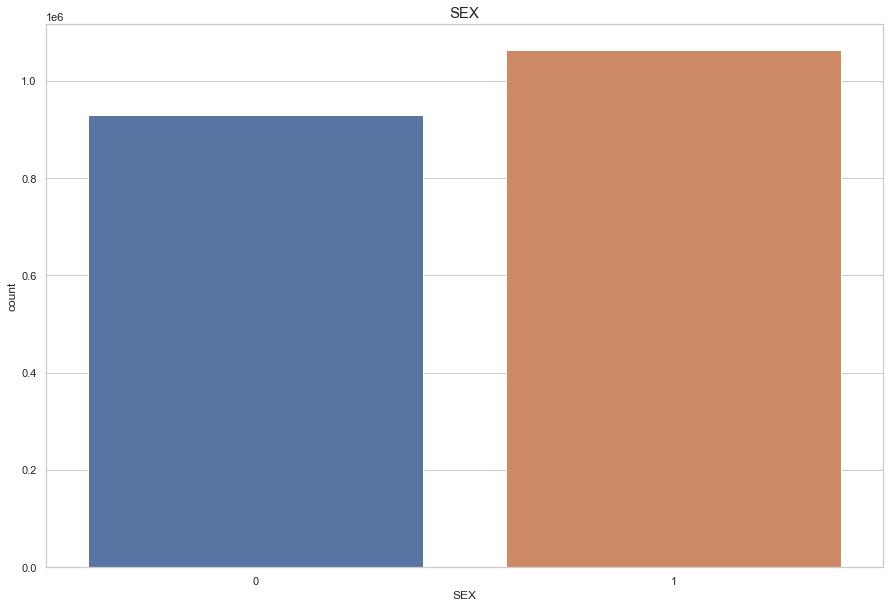
df['SEX'].replace({2:0}, inplace=True)
plt.figure(figsize=(15,10))
sns.countplot(df['SEX'])
plt.title("SEX",fontsize=15)
plt.show()
/Users/minsuha/anaconda3/lib/python3.8/site-packages/seaborn/_decorators.py:36: FutureWarning: Pass the following variable as a keyword arg: x. From version 0.12, the only valid positional argument will be `data`, and passing other arguments without an explicit keyword will result in an error or misinterpretation.
warnings.warn(
df[["SEX", "BLDS"]].groupby(['SEX'], as_index=False).mean().sort_values(by='BLDS', ascending=False)
| SEX | BLDS | |
|---|---|---|
| 1 | 1 | 0.093142 |
| 0 | 0 | 0.054500 |
SIDO
결측치 없음
지역별로 당뇨병환자의 차이가 있다.
one-hot-encoding해준다.
df[['SIDO', 'BLDS']].groupby(['SIDO'], as_index=False).mean().sort_values(by='BLDS', ascending=False)
| SIDO | BLDS | |
|---|---|---|
| 13 | 46 | 0.101004 |
| 9 | 42 | 0.084617 |
| 12 | 45 | 0.083721 |
| 1 | 26 | 0.080649 |
| 3 | 28 | 0.078306 |
| 11 | 44 | 0.078168 |
| 14 | 47 | 0.077858 |
| 10 | 43 | 0.077433 |
| 4 | 29 | 0.075702 |
| 15 | 48 | 0.074349 |
| 16 | 50 | 0.074158 |
| 5 | 30 | 0.073061 |
| 8 | 41 | 0.072273 |
| 2 | 27 | 0.069280 |
| 0 | 11 | 0.068835 |
| 6 | 31 | 0.067514 |
| 7 | 36 | 0.059593 |
mapper = {11 : 'seoul',
26 : 'busan',
27 : 'dagu',
28 : 'incheon',
29 : 'kwangju',
30 : 'dajeon',
31 : 'ulsan',
36 : 'sejong',
41 : 'gyeonggi',
42 : 'gangwon',
43 : 'chongB',
44 : 'chongN',
45 : 'jeonB',
46 : 'jeonN',
47 : 'gyeongB',
48 : 'gyeongN',
49 : 'jeju'}
df['SIDO'].replace(mapper, inplace=True)
df['SIDO'] = df['SIDO'].astype('category')
df = pd.get_dummies(df)
HEAR_LEFT & HEAR_RIGHT
수정
- 잘들림 : 1
- 잘안들림 : 0
청력이 안좋을수록 당뇨병 환자일 확률이 놓으므로 결측치를
- 당뇨병환자 -> 청력 0
- 정상인 -> 청력 1
로 채워준다
df['HEAR_LEFT'].replace({2 : 0}, inplace=True)
df['HEAR_RIGHT'].replace({2 : 0}, inplace=True)
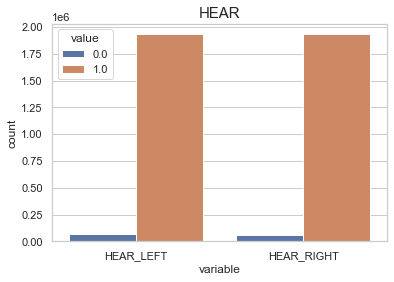
print(df['HEAR_LEFT'].value_counts())
print(df['HEAR_RIGHT'].value_counts())
1.0 1929280
0.0 64406
Name: HEAR_LEFT, dtype: int64
1.0 1931611
0.0 62077
Name: HEAR_RIGHT, dtype: int64
df[["HEAR_LEFT", "BLDS"]].groupby(['HEAR_LEFT'], as_index=False).mean().sort_values(by='BLDS', ascending=False)
| HEAR_LEFT | BLDS | |
|---|---|---|
| 0 | 0.0 | 0.126820 |
| 1 | 1.0 | 0.073391 |
df[["HEAR_RIGHT", "BLDS"]].groupby(['HEAR_RIGHT'], as_index=False).mean().sort_values(by='BLDS', ascending=False)
| HEAR_RIGHT | BLDS | |
|---|---|---|
| 0 | 0.0 | 0.126440 |
| 1 | 1.0 | 0.073468 |
c1 = (df['BLDS'] == 1) & (df['HEAR_RIGHT'].isnull()) #fill 0
c2 = (df['BLDS'] == 0) & (df['HEAR_RIGHT'].isnull()) # fill 1
c3 = (df['BLDS'] == 1) & (df['HEAR_LEFT'].isnull()) # fill 0
c4 = (df['BLDS'] == 0) & (df['HEAR_LEFT'].isnull()) # fill 1
df.loc[c1,'HEAR_RIGHT'] = df.loc[c1,'HEAR_RIGHT'].fillna(0)
df.loc[c2,'HEAR_RIGHT'] = df.loc[c2,'HEAR_RIGHT'].fillna(1)
df.loc[c3,'HEAR_LEFT'] = df.loc[c3,'HEAR_LEFT'].fillna(0)
df.loc[c4,'HEAR_LEFT'] = df.loc[c4,'HEAR_LEFT'].fillna(1)
sns.countplot(x="variable", hue="value", data=pd.melt(df[['HEAR_LEFT', 'HEAR_RIGHT']]))
plt.title("HEAR",fontsize=15)
plt.show()
SMK_STAT_TYPE_CD
수정
- 비흡연, 금연 : 1
- 흡연 : 0
흡연율하는 사람이 당뇨병일 확률이 근소하게 높으므로 378개의 결측치를
당뇨병환자 -> 흡연 0
정상인 -> 금연 1
로 채워준다
df['SMK_STAT_TYPE_CD'].replace({2:1}, inplace=True)
df['SMK_STAT_TYPE_CD'].replace({3:0}, inplace=True)
df[["SMK_STAT_TYPE_CD", "BLDS"]].groupby(['SMK_STAT_TYPE_CD'], as_index=False).mean().sort_values(by='BLDS', ascending=False)
| SMK_STAT_TYPE_CD | BLDS | |
|---|---|---|
| 0 | 0.0 | 0.091915 |
| 1 | 1.0 | 0.070516 |
c1 = (df['BLDS'] == 1) & (df['SMK_STAT_TYPE_CD'].isnull()) #fill 0
c2 = (df['BLDS'] == 0) & (df['SMK_STAT_TYPE_CD'].isnull()) # fill 1
df.loc[c1,'SMK_STAT_TYPE_CD'] = df.loc[c1,'SMK_STAT_TYPE_CD'].fillna(0)
df.loc[c2,'SMK_STAT_TYPE_CD'] = df.loc[c2,'SMK_STAT_TYPE_CD'].fillna(1)
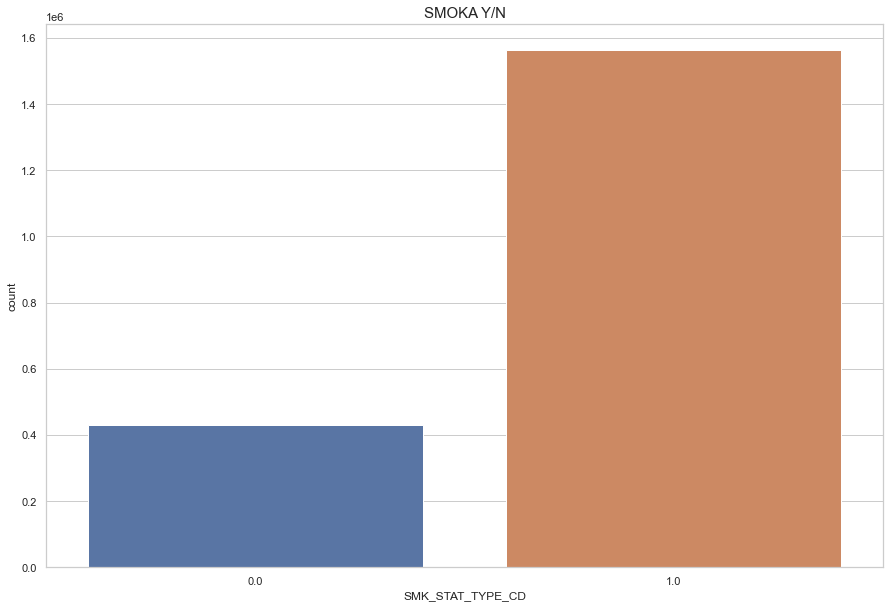
plt.figure(figsize=(15,10))
sns.countplot(df['SMK_STAT_TYPE_CD'])
plt.title("SMOKA Y/N",fontsize=15)
plt.show()
/Users/minsuha/anaconda3/lib/python3.8/site-packages/seaborn/_decorators.py:36: FutureWarning: Pass the following variable as a keyword arg: x. From version 0.12, the only valid positional argument will be `data`, and passing other arguments without an explicit keyword will result in an error or misinterpretation.
warnings.warn(
DRK_YN
수치상으로 음주 여부는 당뇨병과 관련이 없어보이지만, 알코올이 당뇨병을 악화시키므로 당뇨병 환자인 사람은 음주를 하는 사람으로 결측치를 채운다.
df[["DRK_YN", "BLDS"]].groupby(['DRK_YN'], as_index=False).mean().sort_values(by='BLDS', ascending=False)
| DRK_YN | BLDS | |
|---|---|---|
| 0 | 0.0 | 0.075893 |
| 1 | 1.0 | 0.071309 |
c1 = (df['BLDS'] == 1) & (df['DRK_YN'].isnull()) #fill 1
c2 = (df['BLDS'] == 0) & (df['DRK_YN'].isnull()) # fill 0
df.loc[c1,'DRK_YN'] = df.loc[c1,'DRK_YN'].fillna(1)
df.loc[c2,'DRK_YN'] = df.loc[c2,'DRK_YN'].fillna(0)
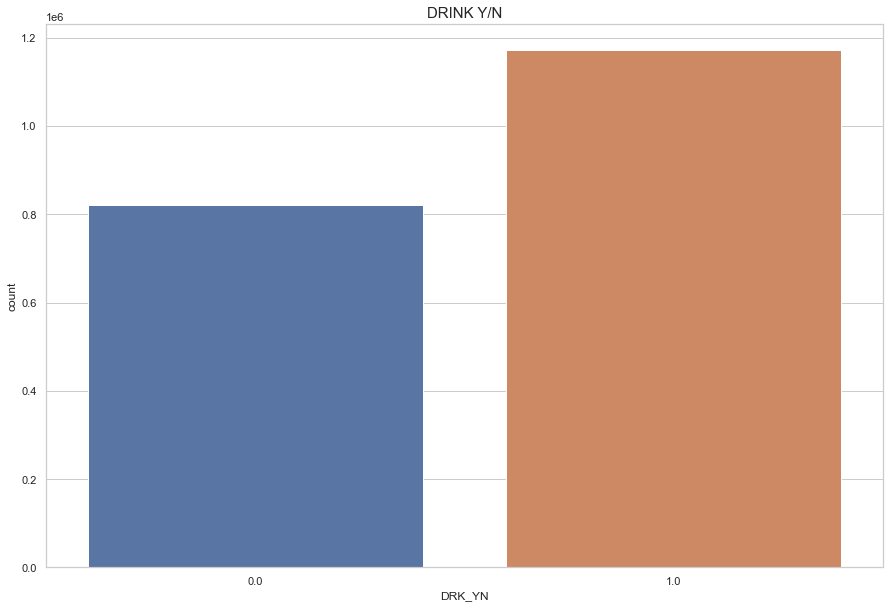
plt.figure(figsize=(15,10))
sns.countplot(df['DRK_YN'])
plt.title("DRINK Y/N",fontsize=15)
plt.show()
/Users/minsuha/anaconda3/lib/python3.8/site-packages/seaborn/_decorators.py:36: FutureWarning: Pass the following variable as a keyword arg: x. From version 0.12, the only valid positional argument will be `data`, and passing other arguments without an explicit keyword will result in an error or misinterpretation.
warnings.warn(
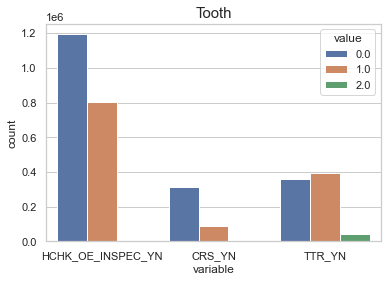
HCHK_OE_INSPEC_YN(구강검진)
구강검진을 받겠다고 한 사람들에 한해서 정보를 얻을 수 있기 때문에 3개의 치아관련 columns들을 같이 살펴본다.
- HCHK_OE_INSPEC_YN(구강검진)
- CRS_YN(치아우식증)
- TTR_YN(치석 유무)
TTR_YN은 HCHK_OE_INSPEC_YN의 1의 개수많큼 있다.
- 구강검진 검사를 받은 사람들에대한 데이터만 존재
치아관련 feature은 결측치가 너무 많으므로 보류
sns.countplot(x="variable", hue="value", data=pd.melt(df[['HCHK_OE_INSPEC_YN', 'CRS_YN', 'TTR_YN']]))
plt.title("Tooth",fontsize=15)
plt.show()
# 구강검진 받은 사람들
sum(df['HCHK_OE_INSPEC_YN'] == 1)
800365
# 치아우식증 결측치아닌 데이터 개수
df['CRS_YN'].notnull().sum()
399860
# 치석 결측치아닌 데이터 개수
df['TTR_YN'].notnull().sum()
800365